Государственный контракт № 02.435.11.1008 |
|
Результаты валидации программы SOL
|
|
Выявление набора активных соединений из массива неактивных Основым результатом работы программы докинга является позиция лиганда в активном центре белка и соответствующее ей значение скоринг функции, характеризующее наибольшую свободную энергию связывания белок-лиганд. Чем более отрицательно это значение скоринг функции, тем лучше лиганд позиционируется в белке. Таким образом, качественно работающая программа докинга должна хорошо отличать активные для данного протеина лиганды от неактивных, выдавая существенно различные значения скоринг функции для обоих типов соединений. То есть, активные лиганды должны обладать значительно более отрицательным значением скоринг функции по сравнению с неактивными (или <мусором>). Таким образом, ранжируя весь набор задоченных лигандов по скорингу, программа докинга должна выводить в верхнюю часть списка лиганды, активные для данного белка, и обладающие более отрицательным значением скоринг функции. Чем больше активных лигандов попадет на самый верх списка, тем более качественно работает программа докинга, позволяя более точно определять потенциально активные лиганды. Это свойство программы докинга можно использовать для компьютерного (виртуального) перебора баз данных химических соединений с целью выявления в них активных лигандов для заданных белков-мишеней, т.е. выявления кандидатов в ингибиторы для заданных белков-мишеней. Такой процесс называется виртуальным скринингом. Итак, для проведения данного типа валидации необходимо выбрать несколько белков-мишеней, сформировать набор неактивных для данных белков лигандов, для каждого из взятых белков-мишеней найти в литературе активные лиганды, т.е. ингибиторы данного белка-мишени, построить 3D-структуры этих активных лигандов и сделать несколько валидационных наборов лигандов (по одному валидационному набору на каждый белок-мишень), каждый из которых содержит неактивные лиганды (<мусор>) и активные для данного белка-мишени лиганды (ингибиторы данного белка-мишени). Для проведения валидации по данному направлению был выбран следующий набор белков-мишеней:
Результаты В ходе проведения валидации было подготовлено 1893 лиганда из базы данных NCI Diversity. Весь этот набор рассматривается как <мусор> для выбранных белков. Соответственно для всех четырех белков проводился докинг всего набора. Для того, чтобы отсортировать <мусор> необходимо провести докинг лигандов активных для данного белка. Для каждого из белков в наборе было выбрано и посчитано:
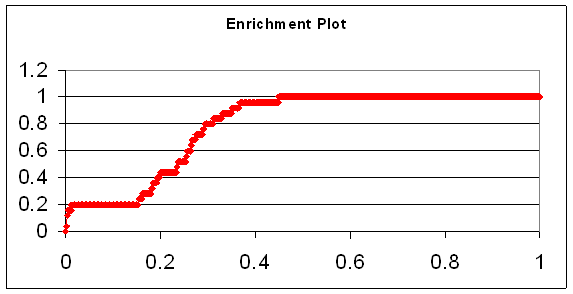
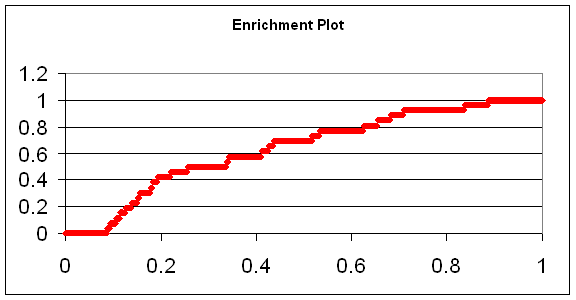
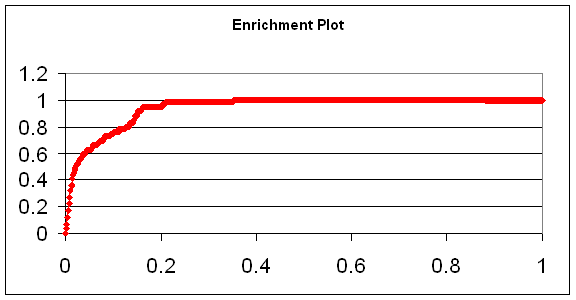
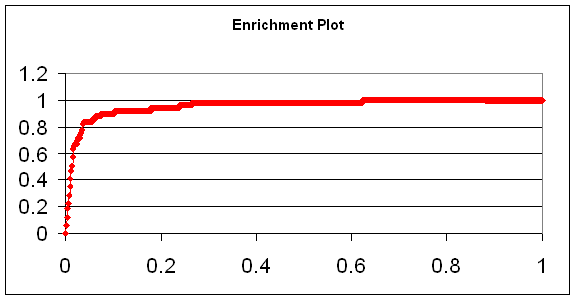
Таким образом, общее количество рассчитанных лигандов для каждого из белков не превышало 2000. При расчете значений enrichment value (EV) мы ориентируемся на следующие критерии качества работы программы докинга, принятые в литературе: значения площади (enrichment value (EV)) 0.6-0.7 - удовлетворительное, 0.7-0.9 - хорошее, > 0.9 - отличное качество работы программы докинга в режиме виртуального скрининга. Приведем графики enrichment plot и значения enrichment value для всех 4 белков-мишеней на полном наборе лигандов, соответственно. Проведение данного типа валидации осуществлялось при следующих параметрах программы докинга SOL. NUMBER OF RUNS: 30 Тромбин представлял собой исключение, в данном случае использовались параметры: NUMBER OF RUNS: 20
Тромбин
p38 MAP киназа
Фактор Xa
Рецептор эстрогена
Один из 4 белков (киназа) попадает в интервал 0.6 - 0.7, что является удовлетворительным качеством скрининга. Результат для тромбин попадает в интервал 0.7 -0.9, что является хорошими качеством скрининга. Для двух других (фактор 10а и рецептор эстрогена) результат превышает 0.9, что является отличным качеством скрининга. В целом на основании этих результатов можно считать, что качество работы программы докинга SOL в режиме виртуального скрининга хорошее. |